Holliday ühendus
Holliday ühendus (ingl. Holliday junction) on DNA nelja ahela vaheline liikuv ühendus, mis ilmub, kui toimub kromosoomide konjugatsioon meioosis.[1] Need ahelad võivad võtta palju erinevaid konformatsioone sõltuvalt soolakontsentratsiooni puhverlahusest ja ümbritsevate nukleotiidide järjestustest, mis asuvad ühenduse lähedal.[2]

Elusrakkudes on Holliday ühendused olulised vahesaadused, mis tekivad geneetilistel protsessidel nagu kaheahelalise katkete rekombinatsiooni ja reparatsiooni tulemusena. Üldjuhul omavad need ühendused sümmeetriliste nukleotiidide järjestusi ning seetõttu omavad teatud mobiilsust, mis tähendab, et neli individuaalset ahelat võivad libiseda läbi struktuuri spetsiifiliselt, suuresti säilitades lämmastikaluste paardumist. Lisaks ilmuvad Holliday ühendusega sarnased neljaahelalised struktuurid mõnedes funktsionaalsetes RNA molekulides.[2]
Liikumatud ja asümmeetriliste järjestustega Holliday ühendused seovad ahelaid teatud asendis, need olid kunstlikult loodud mudeliks, et uurida algupärast Holliday ühendust. Need ühendused leidsid hiljem kasutuse DNA nanotehnoloogias struktuursete ehitusplokkidena, kus mitmeid Holliday ühendusi saab kombineerida ühtseks struktuuriks konkreetse geomeetriaga, moodustades molekule suure struktuurse jäikusega.[3][4]
Struktuur[muuda | muuda lähteteksti]


Holliday ühendused võivad eksisteerida erinevate konformatsiooni isomeeridena erinevate struktuuridega koaksiaalselt pakitud (ingl. coaxial stacking) nelja kaksikahela vahel. Koaksiaalne pakkimine on nukleiinhapete tömbiotsteliste struktuuride kalduvus omavahel seonduda väljapoole avatud lämmastikaluste seondumise teel. On olemas kolm erinevat konformeeri, mis erinevad järjestamisega: vorm, kus puudub koaksiaalne pakkimine ja kaks vormi koaksiaalse pakkimisega. Pakitud vorm valitseb, kui puuduvad kahevalentse metalli katioonid, näiteks Mg2+. Juba vähemalt 0,1 mM Mg2+ juuresolekul neutraliseeritakse elektrostaatiline tõukumine ning domineerivad pakitud struktuurid.[2] Vormid, kus puudub pakkimine, omavad peaaegu lamedat, kandilist struktuuri. Pakkimisega konformeerid koosnevad kahest kaheahelalisest domeenist, mis asuvad 60° nurga all parema käe reegli järgi. Kaks neljast ahelast (üks igast domeenist) säilitavad heeliksstruktuuri ja teised kaks liiguvad ühest domeenist teise antiparalleelsel (ingl. antiparallel) viisil.[2] Kaks võimalikku konformeeri erinevad üksteisest pakkimisega olenevalt sellest, missugustes ahelates on pakkimine toimunud. Ühe vormi ülekaalu määrab konkreetne nukleotiidne järjestus liitumispunkti lähedal. Mõned neist järjestustest on sellised, et kaks konformeeri on üksteisega tasakaalus, samal ajal kui teised järjestused määravad ühe konformeeri ülekaalu. Seega Holliday ühendites, mille nelja ahela ühenduspunktis on järjestus A-CC, on oluliselt ülekaalus konformeer, mis võimaldab vesiniksidemete moodustumist teise tsütosiini ja fosfaatühendi vahel.[2]
Sümmeetriliste järjestustega Holliday ühendite nelja ahela ühenduspunkt (harupunk) saab liikuda juhusliku käigu (ingl. random walk) mudeli järgi. Harupunkti liikumise kiirus varieerub märkimisväärselt olenevalt ioonide kontsentratsioonist: kui ioonide puudumisel ühe akti nihke vahekorra kestus oli 0,3–0,4 ms, siis 10 mM Mg2+ juuresolekul oli see 270–300 ms. Pakkimisega struktuurid asendavad pakkimata struktuure ning see põhjustab kiiruse muutust.[2]
Holliday ühendid RNAst muutuvad antiparalleelseks konformatsiooniks pakkimisega juhul kui magneesiumi kontsentratsioon on kõrge, risti konformatsiooniks pakkimisega kui kontsentratsioon on keskmine ja madala kontsentratsiooni juhul paralleelseks konformatsiooniks; aga isegi madala kaltsiumi kontsentratsioonidega muutuvad nad antiparalleelseks konformatsiooniks.[2]
Bioloogiline funktsioon[muuda | muuda lähteteksti]
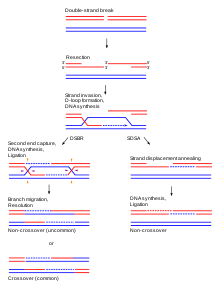
Holliday ühendus on vahevõti homoloogilisel rekombinatsioonil (bioloogiline protsess, mis suurendab geneetilist mitmekesisust nihutades geene kahe kromosoomi vahel) ja samuti kohaspetsiifilisel rekombinatsioonil, mis kaasab integraase. Holliday ühendus on kaasatud kaksikahelaliste katkete reparatsioonis. Lisaks võivad tekkida ristikujulised struktuurid, mis hõlmavad Holliday ühendusi, et leevendada spiraalset pinget sümmeetrilistes järjestustes DNA superspiraalides. Kuigi neljaahelaline järjestus esineb ka funktsionaalsetes RNA molekulides, nagu splaisosomaalses RNAs asU1 ja Tobacco ringspot viiruse (TRSV) juuksenõela ribosüüm, sisaldavad need tavaliselt paardumata nukleotiidi paardunud kaksikahela domeeni vahel ning seega ei loeta neid rangelt võetuna Holliday struktuurideks.[2]
Hollidat ühendus moodustub homoloogilisel rekombinatsioonil identsete või peaaegu identsete järjestuste vahel, mis viib järjestused sümmeetrilisele paigutusele ümber keskse ühenduskoha. See võimaldab harude migratsiooni toimumist kohal, kus järjestused liiguvad läbi ühenduskoha.[2]
Paljud valgud on võimelised ära tundma või moonutama Holliday ühenduse struktuuri. Üks selline klass sisaldab ühendust lahustavaid ensüüme, mis lõhustavad ühendusi, mõnikord järjestuse-spetsiifilisel viisil. Sellised valgud moonutavad ühenduse struktuuri eri viisidel, tõmmates ühendust sageli pakkimata konformatsiooni, lõhustades tsentraalset aluspaari ja/või muutes nurki nelja haru vahel. Teised klassid on harude migratsiooni (ingl. branch migration) valgud, mis suurendavad vahetuse taset suurusjärgu võrra ja saidispetsiifilised rekombinaasid.[2] Prokarüootides jagunevad Holliday ühenduse resolvaasid kahte perekonda: integraasid ja nukleaasid, milles on igaüks struktuurilt sarnane, kuigi nende järjestused ei ole konserveerunud.[5]
Eukarüootides on kaks peamist mudelit, kuidas homoloogiline rekombinatsioon parandab kaheahelalisi katkeid DNAs: topelt-katkete reparatsiooni (DSBR) rada (mõnikord nimetatakse kahekordne Holliday ühenduse mudel) ja sünteesist sõltuv haru sidumise (SDSA) rada.[6]
Resolutsioon[muuda | muuda lähteteksti]
Saccharomyces cerevisiae ehk pagaripärmides saab Holliday ühendusi lagundada nelja erinevat rada pidi, mis moodustavad sisuliselt kõik Holliday ühenduse resolutsioonid in vivo.[7] Rada, kus toimub suurem osa ristsiirdeid S. cerevisiaes ja võimalikult ka imetajatel, kaasab EXO1, MLH1-MLH3 heterodimeeri (nn MutL gamma) ja SGS1 (Bloom sündroomi valgu ortoloog) valgud.[7] Holliday ühendusi seob eelistatult MLH1-MLH3 heterodimeer.[8] See on endonukleaas, mis teeb üheahelalisi katkeid superspiraliseerunud kaksikahelalises DNAs.[8][9] MLH1-MLH3 heterodimeer soodustab ristsiirde rekombinandide moodustumist.[10]
Topeltmutandid, kus on kustutatud nii MLH3 (suur rada) ja MMS4 (vähesel määral), näitasid oluliselt vähenenud ristsiiret võrreldes metsiktüübiga (6–17-kordne erinevus); kuid spooride elujõulisus oli suhteliselt kõrge (62%) ja ilmus peamiselt funktsionaalne kromosoomide lahknemine.[10] Kuigi MUS81 on vähem tähtis pärmi, taimede ja selgroogsete meioosi raja ristsiirde komponent,[11] siis ainuraksel Tetrahymena thermophila näib MUS81 olevat osa olulisest, kui mitte domineerivast ristsiirde rajast.[11] MUS81 osalusega rada on domineeriv ristsiirde mehhanism ka Schizosaccharomyces pombe pärmides.[11]
MSH4 ja MSH5 valgud moodustavad hetero-oligomeerseid struktuure (heterodimeerid) pärmides ja inimestes.[12][13][14] Pärmides soodustab see ristsiiret homoloogiliste kromosoomide vahel meioosis.[12] MSH4/MSH5 kompleks seondub ja stabiliseerib kahekordseid Holliday ühendusi, soodustades nende resolutsiooni ja rekombinantsete ahelate moodustumist. S. cerevisiae mutantidel on osaliselt funktsionaalse MSH4-ga ristsiirete arv genoomi kohta vähendatud 30% võrra ja paljudel juhtudel ei kaasne meioosiga rekombinatsioon. Vaatamata sellele on mutandi spoorid elujõulised, seetõttu toimub homoloogiliste kromosoomide lahknemine õigesti. Järelikult ei ole S. cerevisiae kromosoomi eraldumine meioosis täielikult sõltuv ristsiirdest.[15]
Kasutamine DNA nanotehnoloogias[muuda | muuda lähteteksti]
DNA nanotehnoloogia tegeleb sünteetiliste nukleiinhapete arendamise ja tootmisega, mis ei kanna geneetilist informatsiooni (nagu elusrakkudes), kuid esinevad materjalidena nanotehnoloogias. Hargnenud DNA struktuure kasutatakse põhiühikutena, et luua keerukamaid tehnilisi konstruktsioone. Paljude selliste DNA struktuuride koostisse kuluvad Holliday ühendid. Ühed Holliday ühendid on liiga paindlikud, et neid oleks võimalik koonduda ridadesse, sellepärast kasutatakse jäikade ühikutena struktuurseid motiive, mis koosnevad mitmetest Holliday ühenditest.[3][4] Nendest motiividest levinuim kompleks on topelt ristsiire (DX), mis sisaldab kahte lähestikku paiknevat Holliday ühendit, moodustades seeläbi jäiga struktuuri, mis võib ise koostada kõrgemat järku ridu.[3][4]
Struktuurne motiiv DX on aluskoosteplokk DNA origami meetodis, mida kasutatakse, et luua suuremaid suvalise kujuga kahe- ja kolmemõõtmelisi struktuure.[16]
Kolme kolmnurka kogutud Holliday ühenditest plokke kasutati kolmemõõtmeliste perioodiliste struktuuride loomiseks, mida kasutatakse biomolekulide röntgenanalüüsis.[3][4]
Viited[muuda | muuda lähteteksti]
- ↑ Heinaru. A. (2012). "Geneetika". Tartu Ülikooli kirjastus. lk. 1002.
{{netiviide}}: puuduv või tühi|url=(juhend) - ↑ 2,00 2,01 2,02 2,03 2,04 2,05 2,06 2,07 2,08 2,09 Lilley, David M. J. (2000). "Structures of helical junctions in nucleic acids". Quarterly reviews of biophysics. 33 (2): 109–159. PMID 11131562.
{{netiviide}}: puuduv või tühi|url=(juhend) - ↑ 3,0 3,1 3,2 3,3 Seeman, Nadrian C. (June 2004). "Nanotechnology and the double helix". Scientific American. 290 (6): 64–75 PMID 15195395.
{{netiviide}}: puuduv või tühi|url=(juhend) - ↑ 4,0 4,1 4,2 4,3 Seeman, Nadrian C. (2010). "Nanomaterials based on DNA". Annual Review of Biochemistry. 79: 65–87. PMID 20222824.
{{netiviide}}: puuduv või tühi|url=(juhend) - ↑ Bloomfield, Victor A.; Crothers, Donald M.; Tinoco, Jr., Ignacio (2000). "Nucleic acids: structures, properties, and functions". Sausalito, California: University Science Books.
{{netiviide}}: puuduv või tühi|url=(juhend)CS1 hooldus: mitu nime: autorite loend (link) - ↑ Sung, P; Klein, H. (October 2006). "Mechanism of homologous recombination: mediators and helicases take on regulatory functions". Nature Reviews Molecular Cell Biology. 7 (10): 739–750. PMID 16926856.
{{netiviide}}: puuduv või tühi|url=(juhend)CS1 hooldus: mitu nime: autorite loend (link) - ↑ 7,0 7,1 Zakharyevich, K; Tang, S; Ma, Y; Hunter, N. (Apr 2012). "Delineation of joint molecule resolution pathways in meiosis identifies a crossover-specific resolvase". Cell. 149 (2): 334–347 PMID 22500800.
{{netiviide}}: puuduv või tühi|url=(juhend)CS1 hooldus: mitu nime: autorite loend (link) - ↑ 8,0 8,1 Ranjha L, Anand R, Cejka P. "The Saccharomyces cerevisiae Mlh1-Mlh3 heterodimer is an endonuclease that preferentially binds to Holliday junctions. J Biol Chem. (Feb 2014)". 28;289(9):5674–5686 PMID 24443562.
{{netiviide}}: puuduv või tühi|url=(juhend)CS1 hooldus: mitu nime: autorite loend (link) - ↑ Rogacheva MV, Manhart CM, Chen C, Guarne A, Surtees J, Alani E. (2014). "Mlh1-Mlh3, a meiotic crossover and DNA mismatch repair factor, is a Msh2-Msh3-stimulated endonuclease"". J. Biol. Chem. 289(9): 5664–5673 PMID 24403070.
{{netiviide}}: puuduv või tühi|url=(juhend)CS1 hooldus: mitu nime: autorite loend (link) - ↑ 10,0 10,1 Sonntag Brown M, Lim E, Chen C, Nishant KT, Alani E. (2013). "Genetic analysis of mlh3 mutations reveals interactions between crossover promoting factors during meiosis in baker's yeast". G3: Genes, Genomes, Genetics. 3 (1): 9–22. PMID 23316435.
{{netiviide}}: puuduv või tühi|url=(juhend)CS1 hooldus: mitu nime: autorite loend (link) - ↑ 11,0 11,1 11,2 Lukaszewicz A, Howard-Till RA, Loidl J. (2013). "Mus81 nuclease and Sgs1 helicase are essential for meiotic recombination in a protist lacking a synaptonemal complex". Nucleic Acids Res. 41 (20): 9296–309 PMID 23935123.
{{netiviide}}: puuduv või tühi|url=(juhend)CS1 hooldus: mitu nime: autorite loend (link) - ↑ 12,0 12,1 Pochart P, Woltering D, Hollingsworth NM. (1997). "Conserved properties between functionally distinct MutS homologs in yeast". J. Biol. Chem. 272 (48): 30345–9 PMID 9374523.
{{netiviide}}: puuduv või tühi|url=(juhend)CS1 hooldus: mitu nime: autorite loend (link) - ↑ Winand NJ, Panzer JA, Kolodner RD (1998). "Cloning and characterization of the human and Caenorhabditis elegans homologs of the Saccharomyces cerevisiae MSH5 gene". Genomics. 53 (1): 69–80. PMID 9787078.
{{netiviide}}: puuduv või tühi|url=(juhend)CS1 hooldus: mitu nime: autorite loend (link) - ↑ Bocker T, Barusevicius A, Snowden T, Rasio D, Guerrette S, Robbins D, et al. (1999). "hMSH5: a human MutS homologue that forms a novel heterodimer with hMSH4 and is expressed during spermatogenesis". Cancer Res. 59 (4): 816–22. PMID 10029069.
{{netiviide}}: puuduv või tühi|url=(juhend)CS1 hooldus: mitu nime: autorite loend (link) - ↑ Krishnaprasad GN, Anand MT, Lin G, Tekkedil MM, Steinmetz LM, Nishant KT (2015). "Variation in crossover frequencies perturb crossover assurance without affecting meiotic chromosome segregation in Saccharomyces cerevisiae". Genetics. 199 (2): 399–412. PMID 25467183.
{{netiviide}}: puuduv või tühi|url=(juhend)CS1 hooldus: mitu nime: autorite loend (link) - ↑ Saccà, Barbara; Niemeyer, Christof M. (2012). "DNA Origami: The Art of Folding DNA". Angewandte Chemie International Edition. 51 (1): 58–66. PMID 22162047.
{{netiviide}}: puuduv või tühi|url=(juhend)CS1 hooldus: mitu nime: autorite loend (link)
