Geneetiline assotsiatsiooniuuring
Geneetiline assotsiatsiooniuuring (ingl genome-wide association study, lühend GWAS, varem tuntud ka kui ülegenoomne assotsiatsiooniuuring[1]) on uuringu tüüp geneetikas ja biostatistikas, kus otsitakse seoseid inimese geneetiliste muutujate ja fenotüübi vahel. GWAS-d keskenduvad peamiselt üksiku nukleotiidi polümorfismide ehk SNP-de ja haiguste vahelise seose selgitamisele.
GWAS seisneb kahe uuringugrupi DNA võrdlemises (juhtkontrolluuring). Ühes grupis on haigust põdevad inimesed (juhud) ja teises grupis inimesed, kellel see puudub (kontrollid). Kasutades SNP-de genotüüpimise kiipe (SNP array) uuritakse iga inimese genoomset DNA-d, kasutades kiibil olevaid miljoneid geneetilisi markereid. Kui üht tüüpi SNP-i esineb haigust põdevate inimeste hulgas sagedamini, peetakse antud SNP-i haigusega seotuks. Haigusega seostatud SNP-d loetakse inimese genoomi kindla regiooni markeriks, mis mõjutab riski haigestuda.
GWAS erineb teistest uuringutüüpidest selle poolest, et uuritakse kogu genoomi, mitte ainult ühte või mõnda geneetilist regiooni. Seetõttu on selline mittekandidaatpõhine lähenemine kontrastiks geenspetsiifilistele kandidaatpõhistele uuringutele. Geneetilised assotsisatsiooniuuringud tuvastavad SNP-e ja teisi variatsioone järjestustes, mis võivad olla haigusega seotud olla, aga ainuüksi GWAS põhjal ei saa täpselt öelda, mis geenid on haiguse põhjustajaks ja kuidas need haiguse avaldumises osalevad.[2][3][4]
Ajalugu[muuda | muuda lähteteksti]
Esimene GWAS avaldati 2005. aastal.[5] Seal uuriti kollatähni ealise taandarenguga (age-related macular degeneration, ARMD) patsiente. Selle uuringuga leiti juhtude hulgast kaks SNP-i, millel oli oluliselt erinev alleelisagedus võrreldes kontrollrühmaga.[6]
2012. aastaks oli tehtud üle 1300 GWAS-i, mille käigus on uuritud üle 250 haiguse ja tunnuse, ja avastatud üle 7400 haigustega seostatud SNP-i.[5][7] 2020. aastaks oli tehtud üle 4500 GWAS-i, mille käigus on uuritud üle 5000 haiguse.[5]
Taust[muuda | muuda lähteteksti]

Inimeste genoomide vahel esineb mitmekülgne variatsioon. Võivad esineda nii väikesed ühe nukleotiidi variatsioonid (SNP-d) kui ka suuremad variatsioonid, nagu deletsioonid, insertsioonid ja koopiaarvu variatsioonid (copy number variations, CNV-d). Neist variatsioonidest võib igaüks muuta indiviidi tunnuseid ehk fenotüüpi, näiteks suurendades indiviidi haigusriski või muutes füüsilisi tunnuseid nagu pikkus.[9]
Enne GWAS-ide kasutuselevõttu oli peamiseks uurimismeetodiks perekondades geenide aheldatusel põhinevad pärilikkuse uuringud. Selline lähenemine oli äärmiselt efektiivne ühe geeni põhjustatud haiguste uurimisel, kuid laialt levinud ja komplekshaiguste korral oli keeruline saadud tulemusi korrata.[9][10][11] Ühe võimalusena soovitati geneetilise assotsiatsiooni uuringut. Seda tüüpi uuring uurib, kas teatud geneetilist varianti sisaldav alleel esineb uuritava fenotüübiga inimestes sagedamini, kui on eeldatud. Esmased arvestused näitasid sellise lähenemise suuremat statistilist mõjusust kui seni nõrkade geneetiliste mõjude otsimiseks kasutatud geneetilisel aheleldusel põhinevad uuringud.[12]
GWAS-ide kasutuselevõtule aitasid kaasa biopangad, mis on inimese geneetilise materjali hoidlateks. Biopankade loomine muutis oluliselt odavamaks ja kergemaks uuringute tarbeks vajaliku arvu bioloogiliste proovide kogumise.[13]
Oluline oli ka rahvusvaheline HapMap-projekt, mis alates 2003. aastast on tuvastanud enamiku levinud SNP-dest, mida geneetilistes assotsiatsiooniuuringutes kasutatakse.[14] HapMapi projektiga tuvastatud haplogrupi struktuur võimaldas tähelepanu koondada SNP-de alamrühmale, mis kõige rohkem variatsiooni kirjeldab. Oluliseks eelduseks oli ka SNP-de genotüüpimiseks vajalike kiipide areng.[15]
Metoodika[muuda | muuda lähteteksti]
Kõige sagedamini kasutatakse geneetilistes assotsiatsiooniuuringutes juhtkontroll tüüpi uuringut, mis võrdleb kahte suurt gruppi indiviide, kus juhtude näol on tegu indiviididega, kellel on vaadeldav haigus, ja kontrollide puhul tervete indiviididega. Mõlemas grupis olevate indiviidide DNA genotüübitakse tuntud SNP-de suhtes. Kasutatavate SNP-de arv sõltub vastavast uuringust, üldjuhul jääb see arv miljoni lähedale (500 000 – 700 000), mõnel juhul rohkem (kuni 2 500 000 SNP-d). Seejärel uuritakse nende SNP-de alleelsageduste esinemist juhtude ning kontrollide seas.[16]
Sellise uuringu puhul on peamine ühik mõjususe suuruse tõlgendamisel šansside suhe (odds ratio, OR). OR näitab kahe suhtarvu vahelist suhet, mis GWAS-i kontekstis tähendab juhtgrupis nende indiviidide, kellel on spetsiifiline alleel, ja kontrollgrupis olevate indiviidide, kellel on sama alleel, suhtarvude suhet. Kui alleelisagedus juhtgrupis on oluliselt kõrgem kui kontrollgrupis, siis on OR suurem kui 1 ja vastupidi. Lisaks arvutatakse tavaliselt χ2-testi kasutades P-väärtus OR mõjususe määramiseks. OR suhete, mille väärtus erineb tunduvalt 1-st, leidmine ongi geneetiliste assotsiatsiooniuuringu eesmärk, sest see näitab, et SNP on seotud uuritava haigusega.[16]
Juhtkontrolluuringu kasutamisele on mitmeid alternatiive. Üks tavalisematest on kvantitatiivsete fenotüüpiliste andmete, näiteks pikkuse või biomarkerite kontsentratsioonide või isegi geeniekspressiooni, analüüs. Samamoodi on võimalik kasutada ka dominantse või retsessiivse penetrantsuse mustrite uurimiseks mõeldud alternatiivseid statistikuid.[16] Arvutuste tegemiseks kasutatakse bioinformaatilist tarkvara, nagu PLINK, mis toetavad neid alternatiivseid statistikuid.[17]
Tavaliselt võetakse arvesse ka mitmeid muutujaid, mis võivad saadud tulemusi mõjutada. Sellisteks muutujateks on sugu ja vanus. Lisaks on teada, et geneetilised variatsioonid on seostatud geograafiliste ja ajalooliste populatsioonidega, kus mutatsioonid esimesena tekkisid.[18] Seetõttu peavad uuringud arvestama uuringus osalejate geograafilise ja etnilise päritoluga – seda kontrollitakse populatsiooni stratifikatsiooniga.
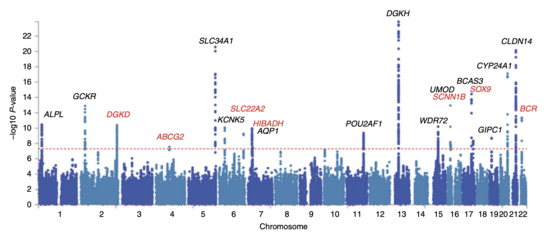
Pärast kõikide SNP-de OR ja p väärtuse arvutamist moodustatakse Manhattani joonis. GWAS-i kontekstis näitab see p väärtuse negatiivset logaritmi genoomil olevate lookuste funktsioonina. Kõige suurema tähtsusega seostatud SNP-d on joonisel selgesti eristatavad punktide virnana haplogrupi struktuuri tõttu. Täpne lävi erineb olenevalt uuringust, aga tavaliselt on p väärtus väga madal (10−7 või 10−8), et seda saaks arvestada kui olulist kõikide testitud SNP-de hulgast. Lisaks kasutatakse tänapäevastes geneetilistes assotsiatsiooniuuringutes metoodikat, kus esimene analüüs tehakse avastatud kohordis, seejärel kontrollitakse tulemusi kõige mõjusamate SNP-de suhtes eraldi tulemuste kontrolliks mõeldud kontrollrühmas.[16]
Tulemused[muuda | muuda lähteteksti]
Geneetiliste assotsiatsiooniuuringute käigus leitud SNP-de, mis on haigusega seotud, tarbeks on püütud luua laialdasi andmebaase.[19] Seoses kiire arenguga GWAS-ide vallas võib leida uusi Vikipeedia stiilis ressursse, mis võimaldaksid luua andmebaase. Hetkel on haigusega seostatud SNP-sid tuhandeid.[7]
Üheks tähtsamaks geneetiliseks assotsiatsiooniuuringuks oli kõige esimene GWAS, mis võrdles 96 patsienti, kellel oli kollatähni ealisuse taandareng, 50 terve kontrolliga. Uuringus leiti kaks SNP-d, mille alleelisagedus kahes grupis oluliselt erines. Need SNP-d leiti komplemendi faktorit H kodeerivast geenist, mis oli ARMD uuringus uudseks avastuseks. Leiud esimesest GWAS-ist on andnud tõuke edasistele funktsionaalsetele uuringutele komplemendisüsteemi terapeutilise manipuleerimise vallas ARMD raviks.[20]
Teine oluline uuring oli 2007. aastal Wellcome Trust Case Control Consortiumi tehtud GWAS, mille käigus uuriti 14 000 juhtu seitsme laiemalt levinud haiguse korral ja 3000 jagatud kontrolli. See oli kõige suurem GWAS, mis uuris aterosklerootilist südamehaigust, esimest tüüpi diabeeti, teist tüüpi diabeeti, reumatoidartriiti, Crohni tõbe, bipolaarset häiret ja hüpertensiooni. Selle uuringuga tuvastati hulgaliselt uusi geene, mis on nende haiguste põhjustajaks.[21][22]
Piirangud[muuda | muuda lähteteksti]
GWAS-ide puhul on kõige tavalistemaks probleemiks hästi määratletud juht- ja kontrollrühmade puudus, liiga väike valim ja populatsiooni stratifikatsiooni puudumine.[3] Üheks probleemiks GWAS-i kui uuringumeetodi puhul on paljude statistiliste testide tegemine, mis toob kaasa suure hulga valepositiivsete tulemuste avaldumise.[3] Korraliku kvaliteedikontrolli ja metoodikaga on neid võimalik vältida.
Lisaks välditavatele probleemidele on genoomiülestes uuringutes osutatud nende laialivalguvusele. GWAS-id uurivad seoste leidmiseks kogu genoomi, mitte lühemaid kandidaatregioone. See on tinginud palju kriitikat GWAS-ide suunas teemal, et GWAS-ide teostamine pole selliseid kulutusi väärt.[23]
Moodsatel GWAS-idel, mis hõlmavad suuri kohorte, on suur võime detekteerida haigusele vastuvõtlikkust mõjutavaid faktoreid ja osutada isegi madalale suhtelisele riskile. Siiski ei ole teada, kas GWAS-i abil on täielikult võimalik mõista komplekshaiguste geneetilist olemust. Limiteerivaks faktoriks on ka koopiaarvu variantide (CNV) laialdane levik tervete inimeste genoomides. CNV-sid leidub küll arvuliselt vähem kui SNP-sid, aga CNV-d hõlmavad mitut nukleotiidi. Nii mõjutavad CNV-d tegelikult suuremat arvu nukleotiidse järjestuse varieeruvust kui SNP-d. Seetõttu on vanema põlvkonna assotsiatsiooniuuringud, mis kasutavad SNP-sid, üsna nõrga võimega detekteerimaks CNV-de poolt põhjustatud vastuvõtlikkust haigusele. Uuema põlvkonna SNP-kiibid (SNP array) sisaldavad siiski juba ka levinumaid CNV-sid.[9]
Vaata ka[muuda | muuda lähteteksti]
Viited[muuda | muuda lähteteksti]
- ↑ "EMS". Eesti märksõnastik. Vaadatud 13.04.2022.
- ↑ Manolio TA; Guttmacher, Alan E.; Manolio, Teri A. (juuli 2010). "Genomewide association studies and assessment of the risk of disease". N. Engl. J. Med. 363 (2): 166–76. DOI:10.1056/NEJMra0905980. PMID 20647212.
- ↑ 3,0 3,1 3,2 Pearson TA, Manolio TA (märts 2008). "How to interpret a genome-wide association study". JAMA. 299 (11): 1335–44. DOI:10.1001/jama.299.11.1335. PMID 18349094.
- ↑ "Genome-Wide Association Studies". National Human Genome Research Institute.
- ↑ 5,0 5,1 5,2 Loos, Ruth (19. november 2020). "15 years of genome-wide association studies and no signs of slowing down". Nature Communications. Vaadatud 13.04.2022.
- ↑ Klein RJ, Zeiss C, Chew EY, Tsai JY, Sackler RS, Haynes C, Henning AK, SanGiovanni JP, Mane SM, Mayne ST, Bracken MB, Ferris FL, Ott J, Barnstable C, Hoh J (aprill 2005). "Complement Factor H Polymorphism in Age-Related Macular Degeneration". Science. 308 (5720): 385–9. DOI:10.1126/science.1109557. PMC 1512523. PMID 15761122.
{{cite journal}}: CS1 hooldus: mitu nime: autorite loend (link) - ↑ 7,0 7,1 Johnson AD, O'Donnell CJ (2009). "An Open Access Database of Genome-wide Association Results". BMC Med. Genet. 10: 6. DOI:10.1186/1471-2350-10-6. PMC 2639349. PMID 19161620.
- ↑ Ikram MK, Sim X, Xueling S; et al. (oktoober 2010). McCarthy, Mark I (toim). "Four novel Loci (19q13, 6q24, 12q24, and 5q14) influence the microcirculation in vivo". PLoS Genet. 6 (10): e1001184. DOI:10.1371/journal.pgen.1001184. PMC 2965750. PMID 21060863.
{{cite journal}}: et al.-i üleliigne kasutus kohas:|author=(juhend)CS1 hooldus: mitu nime: autorite loend (link) - ↑ 9,0 9,1 9,2 Strachan T, Read A. Human Molecular Genetics (4 ed.). Garland Science. Lk 467–495. ISBN 978-0-8153-4149-9.
- ↑ "Online Mendelian Inheritance in Man". Vaadatud 6.12.2011.
- ↑ Altmüller J, Palmer LJ, Fischer G, Scherb H, Wjst M (november 2001). "Genomewide Scans of Complex Human Diseases: True Linkage Is Hard to Find". Am. J. Hum. Genet. 69 (5): 936–50. DOI:10.1086/324069. PMC 1274370. PMID 11565063.
{{cite journal}}: CS1 hooldus: mitu nime: autorite loend (link) - ↑ Risch N, Merikangas K (september 1996). "The future of genetic studies of complex human diseases". Science. 273 (5281): 1516–7. DOI:10.1126/science.273.5281.1516. PMID 8801636.
- ↑ Greely HT (2007). "The uneasy ethical and legal underpinnings of large-scale genomic biobanks". Annu Rev Genomics Hum Genet. 8: 343–64. DOI:10.1146/annurev.genom.7.080505.115721. PMID 17550341.
- ↑ The International HapMap Project, Gibbs RA, Belmont JW, Hardenbol P, Willis TD, Yu F, Yang H, Ch'Ang L-Y, Huang W (detsember 2003). "The International HapMap Project". Nature. 426 (6968): 789–96. DOI:10.1038/nature02168. PMID 14685227.
{{cite journal}}: CS1 hooldus: mitu nime: autorite loend (link) - ↑ Schena M, Shalon D, Davis RW, Brown PO (oktoober 1995). "Quantitative monitoring of gene expression patterns with a complementary DNA microarray". Science. 270 (5235): 467–70. DOI:10.1126/science.270.5235.467. PMID 7569999.
{{cite journal}}: CS1 hooldus: mitu nime: autorite loend (link) - ↑ 16,0 16,1 16,2 16,3 Clarke GM, Anderson CA, Pettersson FH, Cardon LR, Morris AP, Zondervan KT (veebruar 2011). "Basic statistical analysis in genetic case-control studies". Nat Protoc. 6 (2): 121–33. DOI:10.1038/nprot.2010.182. PMC 3154648. PMID 21293453.
{{cite journal}}: CS1 hooldus: mitu nime: autorite loend (link) - ↑ Purcell S, Neale B, Todd-Brown K, Thomas L, Ferreira MA, Bender D, Maller J, Sklar P, de Bakker PI, Daly MJ, Sham PC (september 2007). "PLINK: A Tool Set for Whole-Genome Association and Population-Based Linkage Analyses". Am. J. Hum. Genet. 81 (3): 559–75. DOI:10.1086/519795. PMC 1950838. PMID 17701901.
{{cite journal}}: CS1 hooldus: mitu nime: autorite loend (link) - ↑ Novembre J, Johnson T, Bryc K, Kutalik Z, Boyko AR, Auton A, Indap A, King KS, Bergmann S, Nelson MR, Stephens M, Bustamante CD (november 2008). "Genes mirror geography within Europe". Nature. 456 (7218): 98–101. DOI:10.1038/nature07331. PMC 2735096. PMID 18758442.
{{cite journal}}: CS1 hooldus: mitu nime: autorite loend (link) - ↑ Hindorff LA, Sethupathy P, Junkins HA, Ramos EM, Mehta JP, Collins FS, Manolio TA (juuni 2009). "Potential etiologic and functional implications of genome-wide association loci for human diseases and traits". Proc. Natl. Acad. Sci. U.S.A. 106 (23): 9362–7. DOI:10.1073/pnas.0903103106. PMC 2687147. PMID 19474294.
{{cite journal}}: CS1 hooldus: mitu nime: autorite loend (link) - ↑ Fridkis-Hareli M, Storek M, Mazsaroff I, Risitano AM, Lundberg AS, Horvath CJ, Holers VM (oktoober 2011). "Design and development of TT30, a novel C3d-targeted C3/C5 convertase inhibitor for treatment of human complement alternative pathway–mediated diseases". Blood. 118 (17): 4705–13. DOI:10.1182/blood-2011-06-359646. PMC 3208285. PMID 21860027.
{{cite journal}}: CS1 hooldus: mitu nime: autorite loend (link) - ↑ "Largest ever study of genetics of common diseases published today". Wellcome Trust Case Control Consortium. 6. juuni 2007. Originaali arhiivikoopia seisuga 4.06.2008. Vaadatud 19.06.2008.
- ↑ Wellcome Trust Case Control Consortium, Burton PR, Clayton DG, Cardon LR; et al. (juuni 2007). "Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls". Nature. 447 (7145): 661–78. DOI:10.1038/nature05911. PMC 2719288. PMID 17554300.
{{cite journal}}: et al.-i üleliigne kasutus kohas:|author=(juhend)CS1 hooldus: mitu nime: autorite loend (link) - ↑ Couzin-Frankel J (juuni 2010). "Major heart disease genes prove elusive". Science. 328 (5983): 1220–1. DOI:10.1126/science.328.5983.1220. PMID 20522751.
